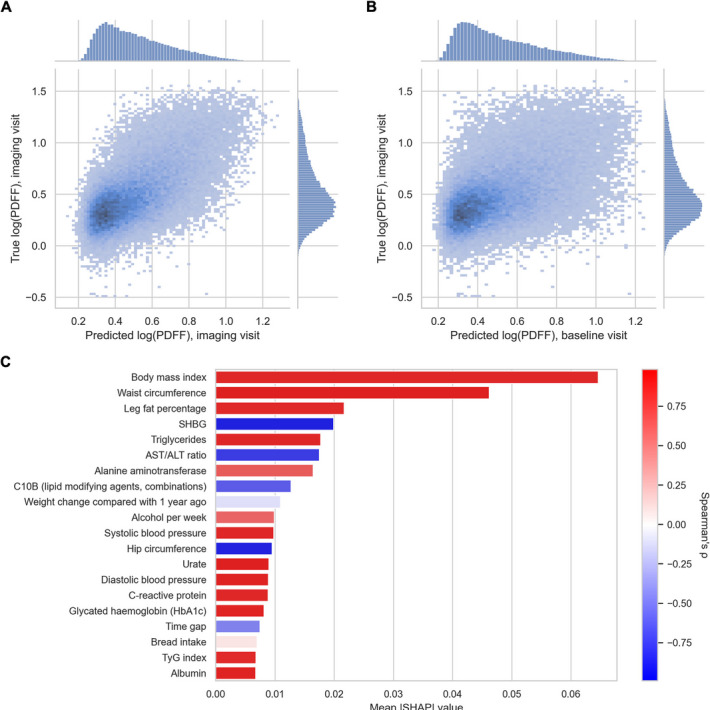
Antecedentes: Los estudios de asociación del genoma completo (GWAS) han identificado variantes comunes asociadas con la enfermedad hepática esteatósica asociada a disfunción metabólica (MASLD). Sin embargo, los estudios de variantes codificantes raras se han visto limitados por las dificultades de fenotipado y el pequeño tamaño muestral. Analizamos las asociaciones de variantes codificantes raras y ultrarraras con la fracción grasa de densidad protónica (PDFF) y el estado de caso-control de MASLD en 736.010 participantes de diversas ascendencias del Biobanco del Reino Unido, All of Us y BioMe, y realizamos un metanálisis transancestral. Posteriormente, desarrollamos modelos para predecir con precisión el estado de PDFF y MASLD en el Biobanco del Reino Unido y analizamos las asociaciones con estos fenotipos predichos para aumentar la potencia estadística.
Resultados: El metaanálisis transancestral con PDFF y estado de caso-control de MASLD identifica dos variantes únicas y dos asociaciones a nivel genético en APOB, CDH5, MYCBP2 y XAB2. Las pruebas de asociación con fenotipos predichos, que replican más variantes genéticas conocidas de GWAS que los fenotipos verdaderos, identifican 16 variantes únicas y 11 asociaciones a nivel genético que implican 23 genes adicionales. Dos variantes fueron polimórficas solo entre los participantes de ascendencia africana y varias asociaciones mostraron heterogeneidad significativa en los análisis de ascendencia y estratificados por sexo. En total, identificamos 27 genes, de los cuales 3 son causas monogénicas de esteatosis (APOB, G6PC1, PPARG), 4 se asociaron previamente con MASLD (APOB, APOC3, INSR, PPARG) y 23 tenían evidencia clínica, experimental y/o genética de apoyo.
Conclusiones: Nuestros resultados sugieren que los análisis de asociación transancestral pueden identificar variantes codificantes raras y ultra raras específicas de la ascendencia en la patogénesis de MASLD. Además, demostramos la utilidad del aprendizaje automático en las investigaciones genéticas de enfermedades difíciles de fenotipificar en biobancos transancestrales.